Práctica 1 (notebook)
Contents
3.1. Práctica 1 (notebook)#
3.1.1. Introducción#
En este notebook se revisan las siguientes funciones:
NetworkX
nx.Graph()nx.draw()G.nodesG.edgesG.degreeG.add_node()G.add_edges()nx.from_numpy_matrix()nx.DiGraph()
Numpy
np.array()np.reshape()A.dot(B)roll
Matplotlib
plt.imshow()
Iniciamos importando las paqueterias necesarias.
import numpy as np
import networkx as nx
import matplotlib.pyplot as plt
3.1.2. Clase Graph()#
La clase Graph() nos permite generar los objetos de mayor interés para este curso: las gráficas o redes. En las siguientes notebooks se explican las funciones más importantes relacionadas con las redes que utilizaremos.
#se crea un objeto de clase Graph y
#se guarda en la variable G para aplicarle funciones y analizar sus propiedades
G = nx.Graph()
# La funcion draw nos da una visualizacion simple de la red generada.
# En este caso, como la red todavia no tiene nodos ni enlaces, solo se
# muestra un "lienzo" blanco:
nx.draw(G)

nx.draw(G)
# Un objeto de tipo Graph tiene una propiedad llamada nodes y es la lista de nodos.
# En el caso de la red que creamos no es otra cosa que una lista sin elementos:
G.nodes
NodeView(())
# Otra propiedad es edges, que es la lista de los enlaces de la red.
# Como se vio antes, un enlace es en primer lugar una pareja de nodos. Entonces,
# la propiedad edges es una lista de parejas de nodos. En el caso de nuestra red
# vuelve a ser una lista sin elementos
G.edges
EdgeView([])
Para un nodo a nuestra red se utiliza la función add_node() . La estructura es [Red].add_node([nombre del nodo]) . El nombre del nodo puede ser un número entero o una cadena de caracteres alfanumericos; en este último caso, se debe escribir entre comillas:
G.add_node('Nodo 1') # se agrega el nodo "Nodo 1"
nx.draw(G) # la visualizacion ya no es un lienzo blanco

G.add_node('Nodo 2') #se agregan otros nodos
G.add_node('Nodo 3')
G.add_node('Nodo 4')
G.add_node('Nodo 5')
nx.draw(G) #se visualiza

Ya que la red tiene varios nodos, se pueden agregar los enlaces mediante la función add_edge(). Notar la estructura de la función que tiene como argumentos los dos nodos que queremos enlazar.
G.add_edge('Nodo 1', 'Nodo 2')
G.add_edge('Nodo 1', 'Nodo 3')
G.add_edge('Nodo 1', 'Nodo 4')
G.add_edge('Nodo 3', 'Nodo 5')
La función draw() puede recibir varios argumentos, además del objeto graph(), como iremos viendo. Para identificar a los enlaces utilizaremos el argumento with_labels = True. Para jugar con el resto de los argumentos de la función, revisar la documentación de la función draw_networkx(), con la que comparte todos los argumentos: https://networkx.org/documentation/networkx-1.7/reference/generated/networkx.drawing.nx_pylab.draw_networkx.html
nx.draw(G, with_labels = True)
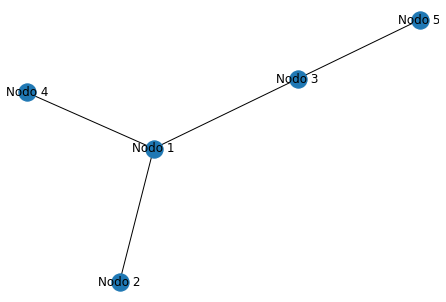
G.nodes
NodeView(('Nodo 1', 'Nodo 2', 'Nodo 3', 'Nodo 4', 'Nodo 5'))
G.edges
EdgeView([('Nodo 1', 'Nodo 2'), ('Nodo 1', 'Nodo 3'), ('Nodo 1', 'Nodo 4'), ('Nodo 3', 'Nodo 5')])
Una última propiedad que se verá por ahora es degree, el grado de los nodos de una red. Se trata de un objeto tipo diccionario en el que a cada nodo le asocia el número «vecinos» que tiene:
G.degree
DegreeView({'Nodo 1': 3, 'Nodo 2': 1, 'Nodo 3': 2, 'Nodo 4': 1, 'Nodo 5': 1})
3.1.3. Estructuras simples#
G1 = nx.Graph()
A la red también se le pueden agregar nodos identificados con números en lugar de cadenas. Esto nos facilitará agregar nodos dentro de bucles:
G1.add_node(0)
G1.add_node(1)
G1.add_node(2)
G1.add_node(3)
G1.nodes
NodeView((0, 1, 2, 3))
for i in range(10):
G1.add_node(i)
nx.draw(G1, with_labels= True)

3.1.3.1. Red lineal#
N = 10
G_lineal = nx.Graph()
for i in range(N - 1):
G_lineal.add_edge(i, i+1)
nx.draw(G1, with_labels = True)

G1.degree
DegreeView({0: 0, 1: 0, 2: 0, 3: 0, 4: 0, 5: 0, 6: 0, 7: 0, 8: 0, 9: 0})
3.1.3.2. Red circular#
G_circular = nx.Graph()
N = 5
for i in range(N):
G_circular.add_node(i)
lista_nodos = list(G_circular.nodes)
for i in range(N):
G_circular.add_edge(lista_nodos[i], lista_nodos[i - 1])
nx.draw(G_circular)
3.1.3.3. Red anillo a segundos, terceros, y n-vecinos#
G_anillo = nx.Graph()
N = 25
for i in range(N):
G_anillo.add_node(i)
lista_nodos = list(G_anillo.nodes)
for i in range(N):
G_anillo.add_edge(lista_nodos[i], lista_nodos[i-1]) #primeros vecinos
G_anillo.add_edge(lista_nodos[i], lista_nodos[i-2]) #segundos vecinos
G_anillo.add_edge(lista_nodos[i], lista_nodos[i-3]) #terceros vecinos
nx.draw_circular(G_anillo)

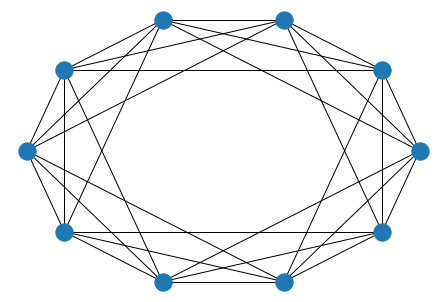
3.1.3.4. Red completa#
G_completa = nx.Graph()
N = 15
for i in range(N):
for j in range(i):
G_completa.add_edge(i,j)
nx.draw_circular(G_completa)
3.1.3.5. Funciones en python#
def Anillo(N):
# generar la red
Red = nx.Graph()
# agregar los nodos
for i in range(N):
Red.add_node(i)
lista_nodos = list(Red.nodes)
for i in range(N):
Red.add_edge( lista_nodos[i-1] , lista_nodos[i] )
Red.add_edge( lista_nodos[i-2] , lista_nodos[i] )
Red.add_edge( lista_nodos[i-3] , lista_nodos[i] )
#graficar la red
nx.draw_circular(Red)
return Red
anillo_20 = Anillo(20)
anillo_20
<networkx.classes.graph.Graph at 0x7f455e5b0a10>
anillo_20.nodes
NodeView((0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19))
anillo_20.edges
EdgeView([(0, 19), (0, 18), (0, 17), (0, 1), (0, 2), (0, 3), (1, 19), (1, 18), (1, 2), (1, 3), (1, 4), (2, 19), (2, 3), (2, 4), (2, 5), (3, 4), (3, 5), (3, 6), (4, 5), (4, 6), (4, 7), (5, 6), (5, 7), (5, 8), (6, 7), (6, 8), (6, 9), (7, 8), (7, 9), (7, 10), (8, 9), (8, 10), (8, 11), (9, 10), (9, 11), (9, 12), (10, 11), (10, 12), (10, 13), (11, 12), (11, 13), (11, 14), (12, 13), (12, 14), (12, 15), (13, 14), (13, 15), (13, 16), (14, 15), (14, 16), (14, 17), (15, 16), (15, 17), (15, 18), (16, 17), (16, 18), (16, 19), (17, 18), (17, 19), (18, 19)])
nx.draw(anillo_20, with_labels = True)
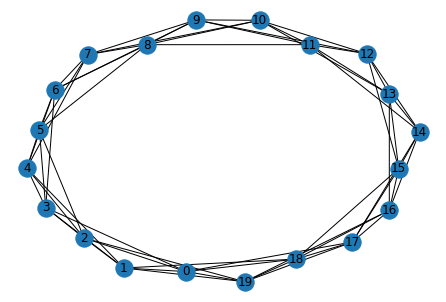
3.1.3.6. Vectorización#
3.1.3.6.1. Red lineal#
N = 20
np.array(range(N))
array([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16,
17, 18, 19])
nodos = np.arange(N)
Para agregar los enlaces de forma vectorial utilizaremos una nueva estructura llamada zip, que intercala los elementos de listas o arreglos. En este caso nos servirá para aparear los nodos, cada uno con el siguiente, mediante el uso de índices de los arreglos. Para ver qué hay dentro de una estructura zip, convirtamosla en una lista:
list(zip(nodos[:-1],nodos[1:]))
[(0, 1),
(1, 2),
(2, 3),
(3, 4),
(4, 5),
(5, 6),
(6, 7),
(7, 8),
(8, 9),
(9, 10),
(10, 11),
(11, 12),
(12, 13),
(13, 14),
(14, 15),
(15, 16),
(16, 17),
(17, 18),
(18, 19)]
Vemos entonces que nos da una lista de parejas de nodos tal y como lo deseamos. Usémoslo ahora para generar la red con estructura lineal
N = 5
nodos = np.arange(N)
red_lineal = nx.Graph()
red_lineal.add_nodes_from(nodos)
red_lineal.add_edges_from( zip(nodos[:-1], nodos[1:] ) )
nx.draw(red_lineal)
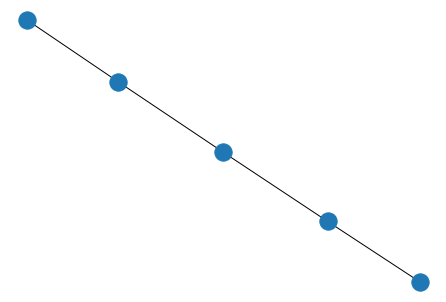
def Red_lineal(N):
nodos = np.arange(N)
red_lineal = nx.Graph()
red_lineal.add_nodes_from(nodos)
red_lineal.add_edges_from( zip(nodos[:-1], nodos[1:] ) )
nx.draw(red_lineal)
return red_lineal
Red_lineal(250)
<networkx.classes.graph.Graph at 0x7f455e55d6d0>

3.1.3.6.2. Red circular#
Para generar la red circular pero ahora de forma vectorial, utilizaremos la función de numpy np.roll()
nodos = np.arange(20)
nodos
array([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16,
17, 18, 19])
np.roll(nodos, 1)
array([19, 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15,
16, 17, 18])
#N = 20
def Red_circular(N):
nodos = np.arange(N)
circular = nx.Graph()
circular.add_nodes_from(nodos)
circular.add_edges_from( zip( nodos, np.roll(nodos, 1) ) )
nx.draw(circular)
return circular
Red_circular(8)
<networkx.classes.graph.Graph at 0x7f455e43c390>
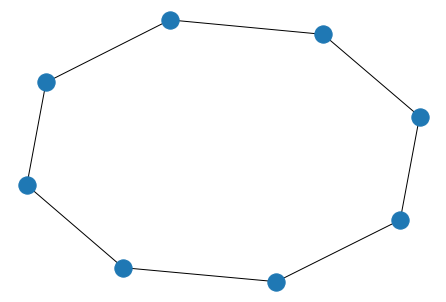
def Red_anillo(N, d):
nodos = np.arange(N)
anillo = nx.Graph()
anillo.add_nodes_from(nodos)
for i in range(d):
anillo.add_edges_from( zip( nodos, np.roll(nodos, i+1) ) )
return anillo
Anillo = Red_anillo(10,3)
nx.draw_circular(Anillo)
3.1.3.6.3. Red completa#
N = 10
completa = nx.Graph()
for i in range(N):
for j in range(i):
completa.add_edge( i , j )
nx.draw_circular(completa)
3.1.4. Arreglos de numpy y matriz de adyacencia#
Ya que comprendemos el formalismo que nos permite pasar de redes a matrices (de adyacencia) y viceversa, podemos emplear funciones de NetworkX que nos permiten lo mismo pero para generar redes (Graph()) de networkx a partir de arreglos de numpy, y viceversa. Esto lo haremos mediante las funciones:
nx.from_numpy_array( )nx.to_numpy_array()
Finalmente, utilizaremos la función plt.imshow() para visualizar las matrices de adyacencia mediante paletas de color que facilitarán el análisis de la estructura de la red.
matriz = [[0,1,1,1],
[1,0,0,0],
[1,0,0,1],
[1,0,1,0]]
A = np.array(matriz)
G = nx.from_numpy_array( A )
nx.draw_circular(G,with_labels = True)
A.dot(A).dot(A)
plt.imshow(A)
<matplotlib.image.AxesImage at 0x7f455c99f2d0>

A = nx.to_numpy_array(G)
A = nx.to_numpy_array(completa)
plt.imshow(A)
<matplotlib.image.AxesImage at 0x7f455c90b910>

red_anillo = Red_anillo(50, 2)
A = nx.to_numpy_array(red_anillo)
plt.imshow(A)
<matplotlib.image.AxesImage at 0x7f76e8ca4f50>

A = nx.to_numpy_array(red_lineal)
plt.imshow(A)
<matplotlib.image.AxesImage at 0x7f76de2c7990>

circular = Red_circular(10)
A = nx.to_numpy_array(circular)
plt.imshow(A)
<matplotlib.image.AxesImage at 0x7f76e8798810>

A = np.array(
[[0,1,1,1,0,1],
[1,0,1,1,0,0],
[1,1,0,0,0,1],
[1,1,0,0,0,0],
[0,0,0,0,0,0],
[1,0,1,0,0,0]
]
)
G = nx.from_numpy_array(A)
nx.draw_circular(G)
Igualmente, como sabemos, las potencias de la matriz de adyacencia proporcionan el número de caminos entre parejas de nodos. Esto puede utilizarse mediante la función dot de numpy, con la cual podemos obtener las potencias de la matriz de adyacencia. A continuación se obtiene el cubo de A, lo que nos daría el número de caminos de longitud tres entre todas las parejas de nodos. Verifica que esta información coincida con la red previamente visualizada (tal vez sean útiles las etiquetas de los nodos).
A.dot(A).dot(A)
3.1.5. Red dirigidas y red con multienlaces#
3.1.5.1. Dirigida (DiGraph)#
G = nx.DiGraph()
G.add_edge('Nodo 1', 'Nodo 5')
G.add_edge('Nodo 1', 'Nodo 4')
G.add_edge('Nodo 2', 'Nodo 3')
G.add_edge('Nodo 3', 'Nodo 5')
G.add_edge('Nodo 2', 'Nodo 5')
nx.draw(G, with_labels = True)
3.1.5.2. Multienlaces (MultiGraph)#
GM = nx.MultiGraph()
GM.add_edge(0,1)
GM.add_edge(0,2)
GM.add_edge(0,3)
GM.add_edge(0,1)
GM.add_edge(0,1)
GM.add_edge(0,1)

